讲了一个什么故事
- 基于人工智能的多模态融合数字病理学和转录组特征能够提高癌症诊断(分级/分型)和预后(生存风险)预测。
- 组织病理学仍然是金标准,而转录组检测在公共卫生中很少被要求。
- 开发了基于扩散模型的跨模态生成式 AI 模型 PathGen,实现从常规组织病理学图像中合成缺失的转录组数据。
- 在四种癌症(胶质瘤、肾癌、子宫癌、乳腺癌)的数据集(TCGA和CPTAC)上进行了严格测试,结果显示:
- 仿真度极高: PathGen合成的基因数据与真实患者的基因数据高度相关(Spearman相关系数显著,特别是原癌基因)。
- 超越单模态: 将“真实的病理图像” + “合成的基因数据”结合使用,其诊断(分级)和预后(生存期)预测的准确率,显著优于仅使用病理图像。
- 媲美真数据:使用“合成基因数据”的模型表现,与使用“真实基因数据”的模型表现在统计学上没有显著差异()。
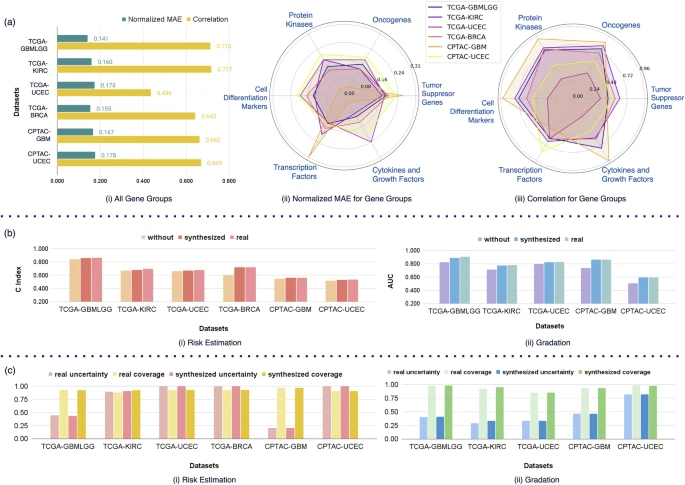
- 可解释性:模型提供了注意力热力图(Attention Maps),并引入了共形预测技术,实现不确定性量化,还还验证了模型在不同年龄、性别群体中的表现。
数据可及性和代码可及性
本文章的数据可及性及代码可及性相当好,代码是开源的,数据用的是公开的 TCGA 和 CPTAC 数据集,可实现完整的结果复现。
- 代码仓库:https://github.com/Samiran-Dey/PathGen
- TCGA 数据集:https://www.cancer.gov/tcga
- CPTAC 数据集:https://gdc.cancer.gov/about-gdc/contributed-genomic-data-cancer-research/clinical-proteomic-tumor-analysis-consortium-cptac
- 转录组相关数据:https://www.cbioportal.org
- 基因列表:https://github.com/mahmoodlab/MCAT/blob/master/datasets_csv_sig/signatures.csv
数据结构
队列
- 胶质母细胞瘤和脑低级别胶质瘤
- TCGA-GBMLGG:745 例 912 张 WSI(183 GBM,562 LGG)
- CPTAC-GBM:62 例 242 张 WSI
- 肾透明细胞癌
- TCGA-KIRC:462 例 485 张 WSI
- 子宫体子宫内膜癌
- TCGA-UCEC:267 例 294 张 WSI
- CPTAC-UCEC:71 例 364 张 WSI
- 乳腺癌
- TCGA-BRCA:946 例 1010 张 WSI
训练集、验证集、测试集的划分:
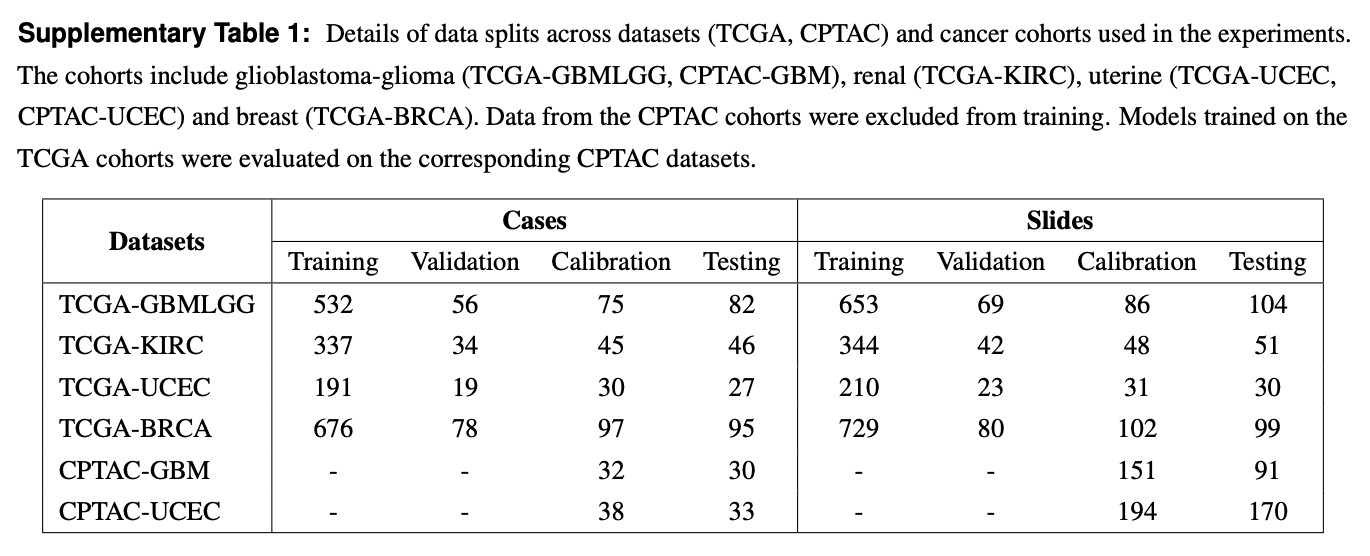
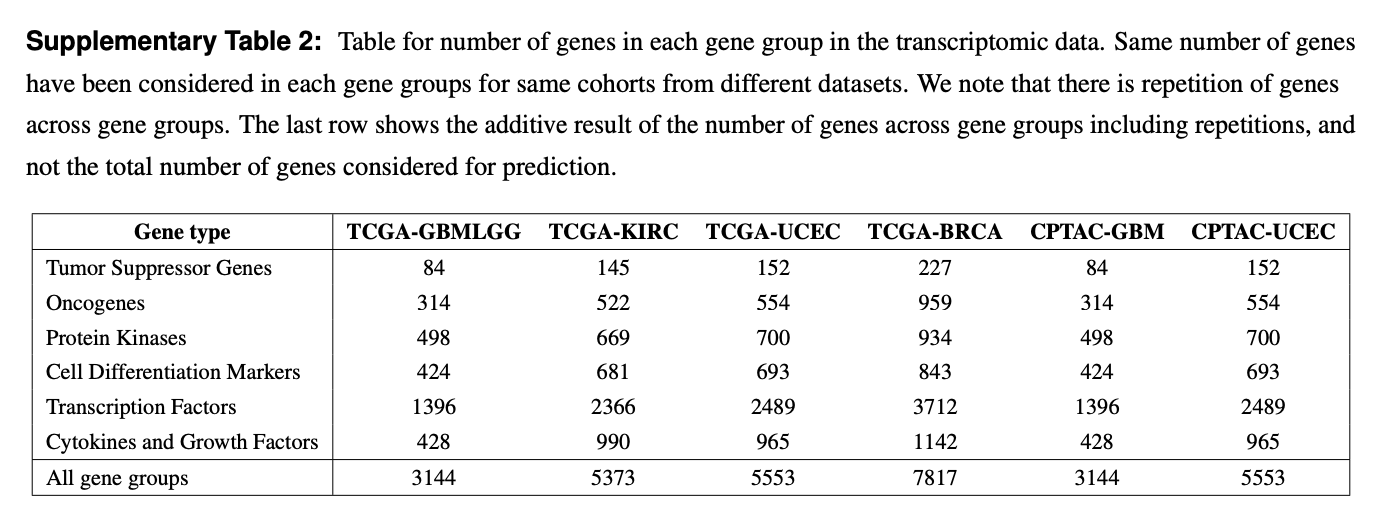
基因组
划分表如下:
Figures
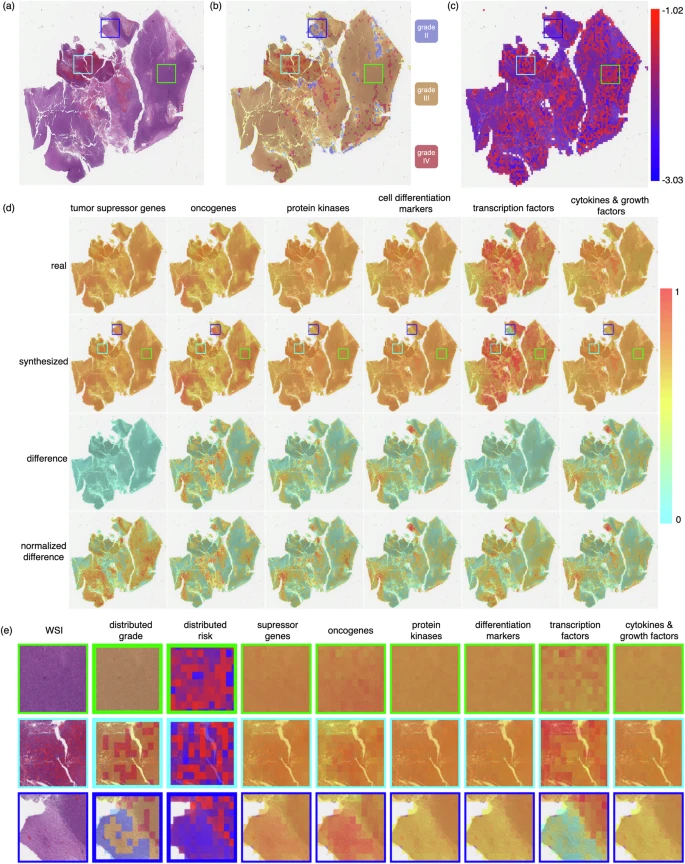
Figure 3: 来自TCGA-LGG队列的一名58岁男性患者的可解释性图谱,其真实分级为III级,生存时间为10.71个月,生存状态为存活。
- a. 全切片图像(WSI)。
- b. 肿瘤内分级异质性。
- c. 肿瘤内生存异质性。
- d. 针对不同基因组,真实与合成转录组数据与WSI图像块之间的协同注意力图。
- e. 对选定区域(以不同颜色矩形标记)使用合成转录组数据进行预测所获得的肿瘤内异质性及其对应协同注意力图的对比研究。
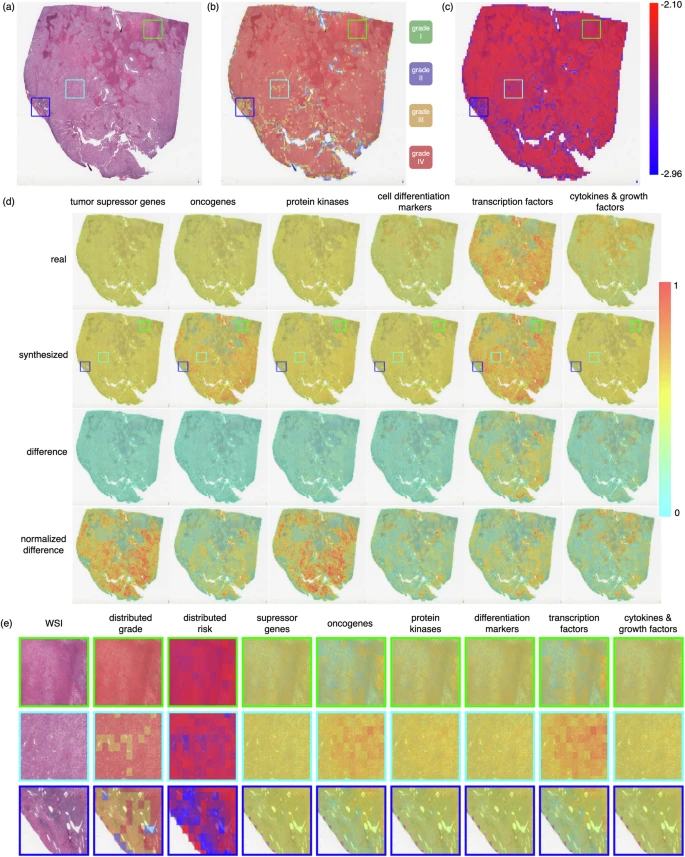
Figure 4: 来自TCGA-KIRC队列的一名40岁男性患者的可解释性图谱,其真实分级为IV级,生存时间为33.99个月,生存状态为死亡。
- a. 全切片图像(WSI)。
- b. 肿瘤内分级异质性。
- c. 肿瘤内生存异质性。
- d. 针对不同基因组,真实与合成转录组数据与WSI图像块之间的协同注意力图。
- e. 对选定区域(以不同颜色矩形标记)使用合成转录组数据进行预测所获得的肿瘤内异质性及其对应协同注意力图的对比研究。
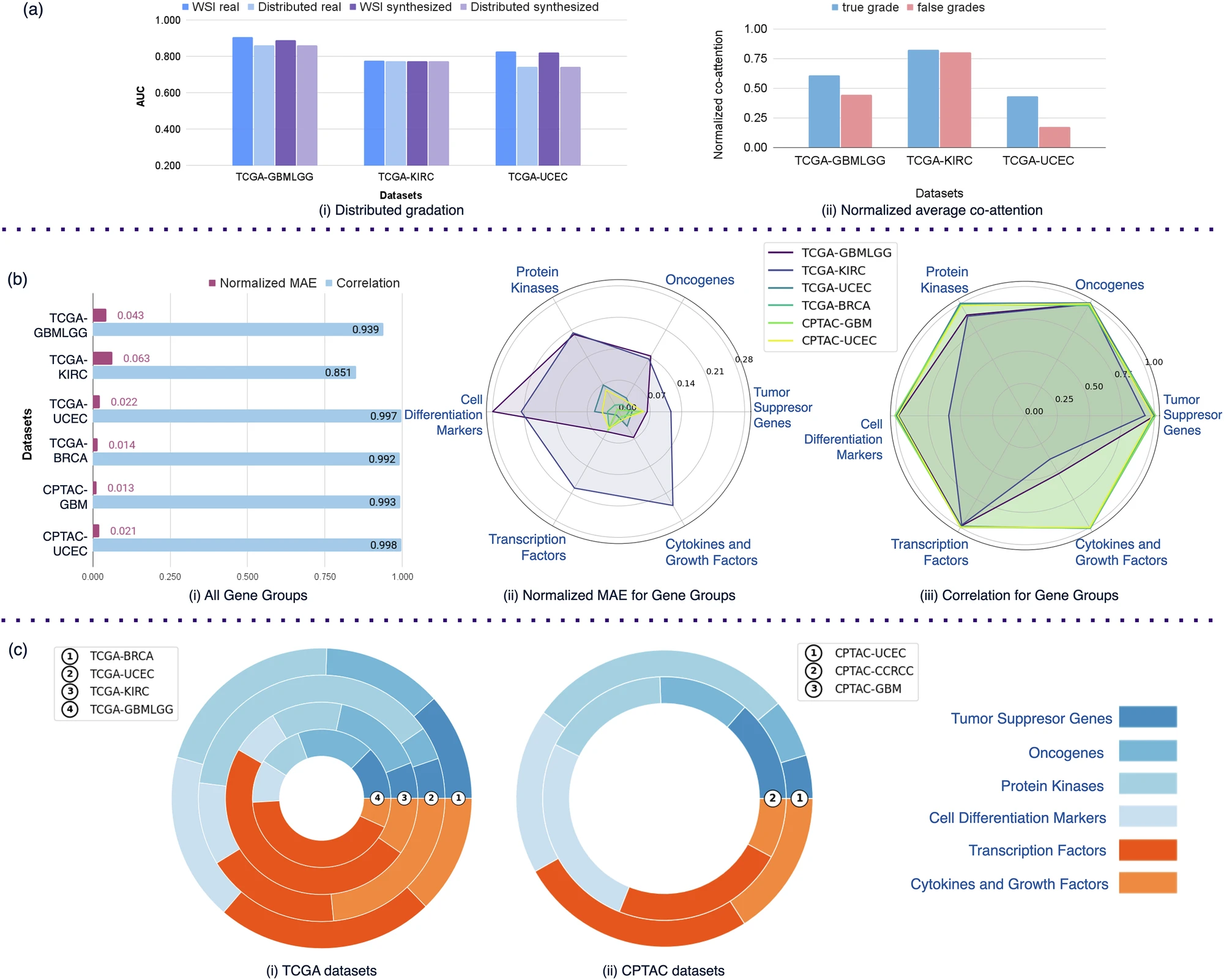
Figure 5: 可解释性分析。
- a. 可解释性分析的对比图。
- (i) 使用真实与合成转录组数据进行分级时,分布式与非分布式全切片图像(WSI)预测结果的对比图;
- (ii) 真实分级的归一化平均协同注意力值高于错误分级的对应值。
- b. 在所有基因及不同基因组上,使用真实与合成转录组数据所得协同注意力图的比较。
- c. 不同TCGA和CPTAC数据队列中各基因组在协同注意力机制中的贡献百分比示意图。源数据以源数据文件形式提供。
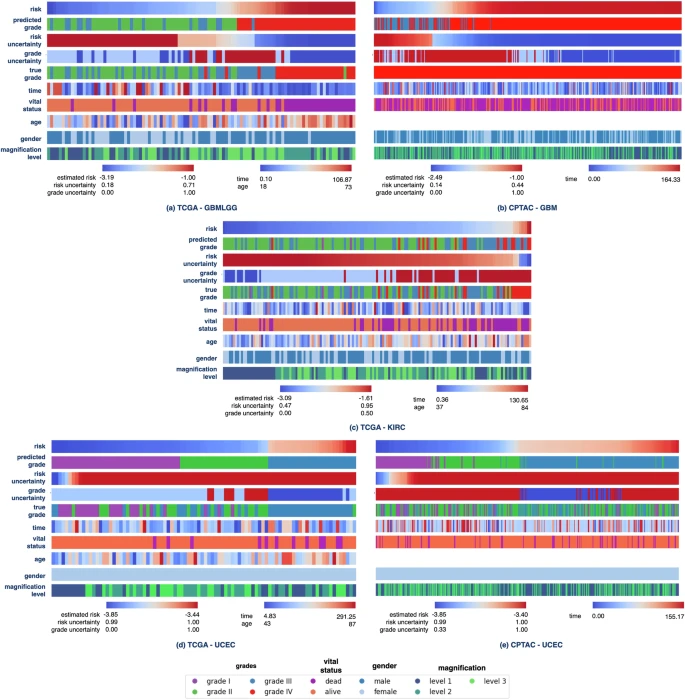
Figure 6: 模型预测在不同人口统计类别和数据队列中公平性评估的示例结果。病例按预测患者风险的升序排列,同时展示了生存风险和分级预测的估计不确定性。源数据以源数据文件形式提供。
代码试运行及结果
详情请见配套文章。